L’intelligenza artificiale per la ricerca sulla struttura delle proteine
Ogni essere umano ha più di 20.000 proteine. Ad esempio l’emoglobina, che si occupa del trasporto dell’ossigeno dai polmoni alle cellule di tutto il corpo, o l’insulina, che indica all’organismo la presenza di zucchero nel sangue.
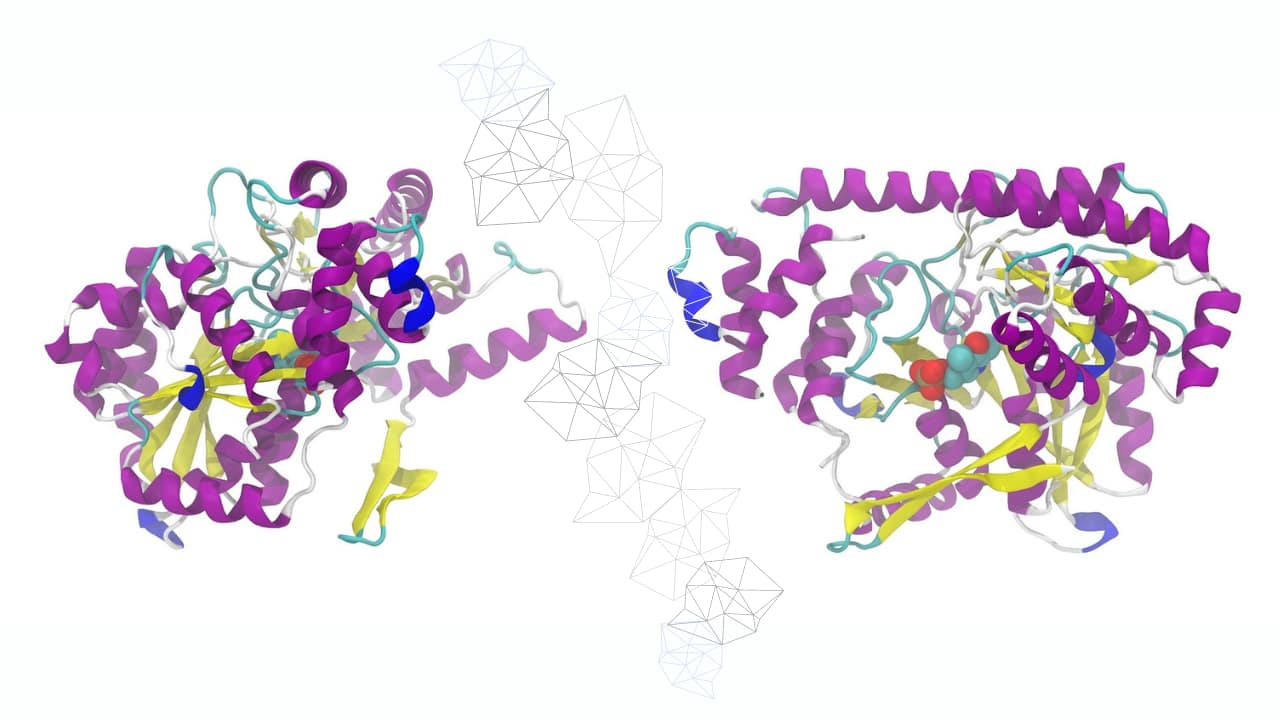
Ogni proteina è costituita da una serie di amminoacidi, la cui sequenza ne determina il ripiegamento e la struttura spaziale – un po’ come se una parola fosse ripiegata nello spazio secondo le sequenze di lettere di cui è composta. Questa sequenza e questo ripiegamento (o struttura) della proteina determinano la sua funzione biologica: il loro studio è il campo della “biologia strutturale”. Si basa su vari metodi sperimentali complementari, che negli ultimi decenni hanno consentito notevoli progressi nella nostra comprensione del mondo vivente, e in particolare consente la progettazione di nuovi farmaci.
A partire dagli anni ’70 si è cercato di conoscere le strutture delle proteine basandosi unicamente sulla conoscenza della sequenza amminoacidica (diciamo “ab initio”). È solo molto di recente, nel 2020, che ciò è diventato possibile in modo quasi sistematico, con l’ascesa dell’intelligenza artificiale e in particolare di AlphaFold, un sistema di intelligenza artificiale sviluppato da un’azienda appartenente a Google.
Di fronte a questi progressi dell’intelligenza artificiale, qual è ora il ruolo dei ricercatori di biologia strutturale?
Per capirlo, bisogna sapere che una delle sfide della biologia di domani è la “biologia integrativa”, che mira a comprendere i processi biologici a livello molecolare nei loro contesti a livello cellulare. Data la complessità dei processi biologici, è essenziale un approccio multidisciplinare. Si basa su tecniche sperimentali, che rimangono essenziali per studiare la struttura delle proteine, la loro dinamica e le loro interazioni. Inoltre, ciascuna delle tecniche sperimentali può beneficiare a modo suo delle previsioni teoriche di AlphaFold.
Indice
Cristallografia a raggi X
La cristallografia è, ad oggi, la tecnica più utilizzata in biologia strutturale. Ha permesso di identificare più di 170.000 strutture proteiche nella “Protein Data Bank“, con più di 10.000 ripiegamenti diversi.
Per usare la cristallografia a raggi X, devi “cristallizzare le proteine“. Si dice spesso che questa tecnica è limitata dalla qualità dei cristalli proteici, che è inferiore per le proteine di grandi dimensioni. Ma non sempre questa nozione corrisponde alla realtà: ad esempio, la struttura del ribosoma, l’enorme macchina molecolare che assembla le proteine, è stata risolta a 2,8 angstrom di risoluzione. Venkatraman Ramakrishnan, Thomas Steitz e Ada Yonath hanno ricevuto il Premio Nobel per la Chimica nel 2009 per questo lavoro.
Con il recente sviluppo del laser a elettroni liberi a raggi X (XFEL), è diventato possibile studiare simultaneamente migliaia di microcristalli proteici a temperatura ambiente e sulla scala del femtosecondo (10-15 secondi, o un milionesimo di miliardesimo di secondo, la scala temporale in cui avvengono le reazioni chimiche e il ripiegamento delle proteine). Questa tecnica rende possibile l’immagine delle proteine prima che vengano distrutte. È in procinto di rivoluzionare la “cristallografia cinetica”, che permette di vedere le proteine “in azione”, così come la ricerca di farmaci.
Per il momento, il contributo di AlphaFold allo studio della struttura proteica mediante cristallografia si è concentrato sulla generazione di modelli proteici sufficientemente precisi da applicare la cosiddetta tecnica della “sostituzione molecolare” alla risoluzione delle strutture.
Spettroscopia di risonanza magnetica nucleare
Un altro metodo sperimentale per studiare la struttura delle proteine è la “spettroscopia di risonanza magnetica nucleare”. Mentre il suo alter ego di imaging medico, la risonanza magnetica, esamina la distribuzione spaziale di un singolo segnale, caratteristico degli elementi chimici nei tessuti biologici osservati, nella spettroscopia di risonanza magnetica nucleare è un insieme di segnali provenienti dagli atomi che costituiscono la proteina che viene registrata (questo è chiamato “spettro”).
Generalmente, la determinazione della struttura della risonanza magnetica è limitata a proteine di dimensioni modeste. I modelli di molecole sono calcolati sulla base di parametri strutturali (come le distanze interatomiche), derivati dall’analisi degli spettri sperimentali. Possiamo immaginarlo come agli inizi della cartografia, dove le distanze tra i punti di riferimento rendevano possibile disegnare mappe 2D. Per facilitare l’interpretazione di spettri che contengono molte informazioni, è possibile utilizzare modelli ottenuti per previsione (piuttosto che sperimentalmente), come con AlphaFold.
Oltre alla determinazione strutturale, la spettroscopia di risonanza magnetica nucleare offre due vantaggi principali. Da un lato, in genere, lo studio viene effettuato con un campione in soluzione acquosa ed è possibile osservare le parti particolarmente flessibili delle proteine, spesso invisibili con altre tecniche. Si può persino quantificare il loro movimento in termini di ampiezza e frequenza, il che è estremamente utile perché la dinamica interna delle proteine è cruciale per la loro funzione quanto la loro struttura.
D’altra parte, la spettroscopia di risonanza magnetica nucleare rende facile rilevare le interazioni delle proteine con piccole molecole (ligandi, inibitori) o altre proteine. Ciò consente di identificare i siti di interazione, informazioni essenziali, tra l’altro, per la progettazione razionale di molecole attive come i farmaci.
Queste proprietà rendono la spettroscopia di risonanza magnetica nucleare uno strumento straordinario per la caratterizzazione funzionale delle proteine in complementarità con altre tecniche sperimentali e AI.
Criomicroscopia elettronica
La criomicroscopia elettronica consiste nel congelamento ultrarapido (circa -180°C) di un campione idratato in un sottile strato di ghiaccio, attraverso il quale passano gli elettroni. Gli elettroni trasmessi genereranno un’immagine del campione che, dopo l’analisi, consente l’accesso a strutture che possono raggiungere una risoluzione atomica. In confronto, un microscopio ottico ha solo un potere risolutivo di poche centinaia di nanometri, che corrisponde alla lunghezza d’onda della luce utilizzata; solo un microscopio che utilizza una sorgente con lunghezze d’onda sufficientemente basse (come gli elettroni per la microscopia elettronica) ha un potere risolutivo teorico dell’ordine di un angstrom. Il Premio Nobel per la Chimica 2017 è stato assegnato a Jacques Dubochet, Richard Henderson e Joachim Frank per il loro contributo allo sviluppo della criomicroscopia elettronica.
Con molti sviluppi tecnologici, compreso quello dei rivelatori di elettroni diretti, dalla metà degli anni 2010, questa tecnica è diventata essenziale nella biologia strutturale avviando una “rivoluzione della risoluzione“. Infatti, la criomicroscopia elettronica consente oggi di ottenere strutture con risoluzione atomica, come nel caso dell’apoferritina – una proteina dell’intestino tenue che contribuisce all’assorbimento del ferro – a 1,25 Angstrom di risoluzione.
Il suo vantaggio principale è quello di consentire di determinare la struttura di oggetti di medie dimensioni, oltre 50.000 Dalton (un Dalton corrisponde approssimativamente alla massa di un atomo di idrogeno), come l’emoglobina di 64.000 Dalton, ma anche oggetti di qualche miliardo di Dalton (come il mimivirus, un virus gigante di circa 0,5 micrometri).
Nonostante tutti i progressi tecnologici sopra menzionati, la criomicroscopia non sempre consente di risolvere con una risoluzione sufficientemente elevata la struttura dei “complessi”, costituiti da più proteine. È qui che AlphaFold può aiutare e permettere, in complementarità con la criomicroscopia, di descrivere le interazioni a livello atomico tra i diversi costituenti di un complesso. Questa complementarità dà nuova forza alla criomicroscopia elettronica per il suo ruolo futuro nella biologia strutturale.
I contributi di AlphaFold
AlphaFold consente di prevedere la struttura delle proteine esclusivamente dalla loro sequenza con le conoscenze acquisite dalla biologia strutturale sperimentale. Questo approccio è rivoluzionario perché le sequenze di molte proteine sono note attraverso gli sforzi di sequenziamento del genoma, ma determinarne sperimentalmente le strutture richiederebbe enormi risorse umane e tecniche.
Allo stato attuale, questo tipo di programma rappresenta quindi un ulteriore attore di complementarità, ma non sostituisce le tecniche sperimentali che, come abbiamo visto, forniscono anche informazioni aggiuntive (dinamica, interfacce), a diverse scale (dai siti metallici ai complessi multiproteici) e più affidabile, perché verificata sperimentalmente. Al di là della pura determinazione strutturale di una proteina isolata, la complessità dei sistemi biologici richiede spesso un approccio multidisciplinare per chiarire i meccanismi e le funzioni di queste affascinanti biomolecole che sono le proteine.
Autore
Beate Bersch, Emmanuelle Neumann, Juan Fontecilla, Grenoble Alpes University (UGA)